JBC論文の画像に対する指摘に関して
平成27年1月2日にWEB上で匿名の人物から、以下の論文のFigure 4Aの画像が不自然であるとのご指摘をいただきました。当該論文を見直しましたところ、ご指摘通りの不適切な部分がありましたので、約16年前の実験ノートとフィルム(生データ)を元に自己点検しました。結果を報告させていただきます。
指摘された論文
Sp1 and NF-Y synergistically mediate the effect of vitamin D(3) in the p27(Kip1) gene promoter that lacks vitamin D response elements.
Inoue T, Kamiyama J, Sakai T.
J Biol Chem. 1999 Nov 5;274(45):32309-17
不自然との指摘をいただいた2件
Fig. 4 A;EMSAゲル写真のレーン1を左右反転するとレーン15と類似している件。
Fig. 4 A;EMSAゲル写真のレーン12/13を左右反転するとレーン16/17と類似している件。
自己点検結果
筆頭著者で、当時当教室に在籍していた井上敏昭助手(現在は他大学准教授)が、16年前の実験ノートとレントゲンフィルムとBASデジタルデータを当教室から他大学への転出後は自宅に保管していました。生データであるレントゲンフィルムとBASデジタルデータを用いて自己点検した結果、以下に詳細に経緯を述べておりますような取り違えであったことが明らかとなりましたので、ここに世界中のアカデミック研究者ソサエティーの方々、および文部科学省をはじめとするアカデミック研究を御支援御指導いただいているご関係の方々にご報告するとともに、お詫びを申しあげます。
また、平成27年2月に大学の予備調査をうけて、実験ノートなど当時の資料を見せて説明させていただき、不正は無かったとの予備調査結果をいただきました。なお、これらの結果を受け、既にJournal of Biological Chemistryにcorrectionを申し入れました。
反省点は以下の2点です。
反省点1:
今回の私達のケースでは、同時に実験に供した複数のゲルから得られた結果を、わかりやすく示すために一つの図に再構成しましたが、その際に2件の取り違えをいたしました。注意が不十分でこのような間違いをおこしたことをお詫び致します。また、そのミスに気づかなかったチェック体制の不備につきましてお詫び申しあげます。
反省点2:
今回の私達のケースでは、同時に実験に供した複数のゲルから得られた結果を、わかりやすく示すために一つの図に再構成しています。また逆に一つの実験をわかりやすく説明するために、複数の図に分けて示すケースもあります。現在のガイドラインの考え方では、そのような場合には、レーン間で切った箇所と異なるゲルからの箇所に数ミリの空白を空けることが求められていますが、当該論文を書いた16年前は空白を空けない示し方を可とする考え方もありましたので当該論文でそのようにしていました。また、再構成などが不要な図を得られるような再実験を行っていればこのような事態が避けられたはずです。それを実行しなかったことを反省しています。今後は過誤や誤解を招かない図が得られるような実験デザインとレーン割り付けで実験を行うことを心がけてまいります。
以上をご報告申しあげますとともに、お詫びを申しあげます。今後は再発防止の努力をして精進していきたいと考えております。
平成27年4月6日
責任著者 酒井敏行
実験ノートと生データを参照する自己点検結果
平成27年3月18日
筆頭著者 井上敏昭
はじめに
Figure 4Aにつきまして、この実験は筆頭著者の井上が行ったものですが、間違った位置のバンドを取り込んでいました。データを慎重に扱えておらず2件の間違いをおこしたことをお詫び申し上げます。また、過誤や誤解を招かない図が得られるような実験デザインとレーン割り付けで実験を行うべきであったと反省しています。
論文の概要とFigure 4の内容
この論文では、Vitamin D3によるp27プロモーター活性化には、その-555/-512の位置にあるSp1結合サイト(二つあるうち、より上流にあるもの)とNF-Y結合サイトが重要な役割をすることを報告しました。
Figure 4Aでは、-555/-512をプローブとしてVitamin D3処理U937細胞の核タンパク質を用いてゲルシフトアッセイを行っています。-555/-512の放射性標識オリゴプローブを使用しましたが、これはSp1結合サイトとNF-Y結合サイトへのタンパク分子の結合が検出されることを期待してのものでした。実際のFigure 4AのデータはSp1結合サイトへのタンパク分子(Figure 4BではこれがSp1であることを示します)の結合のみが見られ、このプローブにおけるNF-Y結合サイトにタンパクは直接結合しないことを示しています。なおSp1結合サイトを含まない、より短いp27プロモータープローブを用いた場合には、そのNF-Y結合サイトにNF-Yが結合しうることはFigure 5で示しています。
Figure 4Aの実施内容とデータ取り込み法
ここではcompetition assayを行っております。これはプローブと核タンパク質との結合反応時に未標識オリゴDNAをプローブに対し過剰量を加え、放射性プローブとタンパクとの結合がcompeteされるかどうか知るassayです。結果としては、その-555/-512の上流側のSp1結合サイトを持つcompetitorで特異的にcompeteされていることから、このサイトへの特異的タンパク結合があることを示します。
この実験手技とデータの取り込みですが、泳動後のアクリルアミドゲルをろ紙に移しとり、ゲルを乾燥させます。その放射性シグナルを当時共同実験機器として設置されていた富士フイルム製イメージング機器BASでdigital画像データとして取り込みました。BAS機器は、X-rayフィルムに比べると画像解像度は劣りますが、検出感度が良いために数十分で結果を早く知ることができ、また定量性においても優れています。ただし解像度が劣ると言っても当時の論文に必要な品質は十分満たしており、また得られるファイルサイズが小さく、当時のPCでも十分扱えるという利点がありました。
BASで解析した乾燥ゲルは、その後X-rayフィルムで数日間増感紙ととともに、-80度で露光し現像して保存します。これにより生データとして保存することもできる上、粒子が細かいよりきれいなデータを保存できます。
取り込み間違いとなった原因の推察
論文に使用した図がそれぞれどの画像から由来したのか、どう間違って取り込んでしまったのかを、BAS画像、オリジナルデータであるX-rayフィルム画像と比較しながらまとめたのが添付の図1です。この図1を元に間違いの原因を推察します。
①ゲルをろ紙に移しとりBAS画像として取り込む際に予想される間違い
ゲルシフトアッセイでは、ゲルを取り出す際には、ガラス板を外し、ゲルをろ紙に移しとります。これは、ゲルは濃度4%と柔らかく単独で取り出すことができないためで、ガラス板のどちらか片側についたゲルにろ紙を押さえつけることで移しとります。このとき、どちらのゲル板に残るかはゲル板を外してみるまではわからず、ろ紙を下にしてみた場合ゲルが正面に向くとは限らず左右反対となることも多くあります。そのためろ紙の片隅に筆記用具で左右上下がわかるようマーキングをします。自分で向きがわかるようどちらか端のレーンにはブロモフェノールブルーを入れるので、マーキングで間違えることは回避できます。この左右のマーキングはX-rayフィルムに現像する際には、自分で向きを認識して手動で現像し、現像後直ちにフィルムに左右の情報を書き込むことができます。しかし、BASで取り込む際には放射性シグナル以外は取り込まれないので、共同機器室にあるBASからdigital画像データとして取り出したのち(Tiff形式だったかと思います)、ゲルの位置情報を元にデスクに持ち帰ってPCで必要に応じ画像ソフトで左右反転を行います。取り込み間違いの大きな原因はこの操作にあったとのではないかと思います。
BAS画像は、digital画像データだけでなくBAS機器付属のピクトログラフィーでそのまま印刷したものも保管しており、そこからは三枚のゲルともBAS画面上では左右反対であり(左右反対でろ紙に移しとらざるを得なかった)、正しい図の表示には左右反転が必要だったことがわかりました。Figure作成時には、左右反転させた画像データと、させる前のデータの双方をPCモニターに同時に出してしまい、どちらが本来の画像であるのかについて途中から認識不十分なまま、それぞれをシグナルのパターンでのみ判断してsplitしてFigureを作成したのだと思います。とくに、今見ると、ゲルの一枚目と二枚目のデータがお互い左右反転するとパターンが似ているので、図のE, F, Gに相当する部分を取り込む際に、それぞれの正しい左右の状態をお互いに見誤ったのではないかと思います。実際パターンが他の二枚と異なる三枚目のゲルではそのような間違いは起きておりません。
②必要最小限のデータでわかりやすく結果を伝えるためにsplitした際に起こる間違い
元のデータにある通り、competitionで加えた未標識のオリゴDNAは実際にはプローブに対して、5, 15, 45倍と量をふって行っていますが、Figure作成の際には見やすく簡略化するために5倍量のレーンはすべて削除しました。さらに上流側のSp1結合サイトにタンパクが結合していること、このプローブでのNF-Y結合サイトにはタンパクが結合しないことをわかりやすく伝えるために、順番を変更しています。これらのことも間違った取り込みの原因になったものと思います。どれも似たバンドが多い上、粒子の粗いBAS画像でのパターン認識を自分で間違えたのではないかと思います。competitorなしのレーン(H:最左)についても、一枚目の2レーン目を切り取ろうとした際に、パターンが酷似する、使用しないはずのバンドと間違って取り込んだのだと思います。
③X-rayフィルムを直接取り込めなかったため
X-rayフィルムにはサンプル名も直接書き込みできるので、今なら文字情報を含めフィルムごと光学スキャナーで取り込めばこのような間違いを減らせます。しかし、X-rayフィルム全体をスキャナーにて高解像度で取り込もうとすると、当時の通常のPCではメモリ不足のアラートが出て、作動しないことが多くありました。また当時使用していたスキャナーではポジフィルムを鮮明、精細にスキャンすることができませんでしたので、あまり使用しませんでした。今ならポジフィルムを鮮明に取り込む透過型スキャナーを利用することになりますが、当時はまだあまり普及していなかったように思います。実際こういった理由から、当時の当研究室をはじめ多くの研究室ではBAS画像を主に使っておりました。
結論は合っているとはいえ、また上記のような理由はあったとしても、その根幹となるデータについて細部を見落とし、間違ったレーンを取り込んで使用し、疑義を生じさせてしまったことをお詫びします。今後はこのような間違いの無いよう肝に銘じます。
Correctionについて
今回、J. Biol. Chem.にcorrectionを依頼しました。その際には、当時のX-rayフィルム全体を透過型スキャナーにて高解像度で取り込みました。原図と同じ配置にしたものに加えてsplitも並び替えも新たに用意しました(図2)。
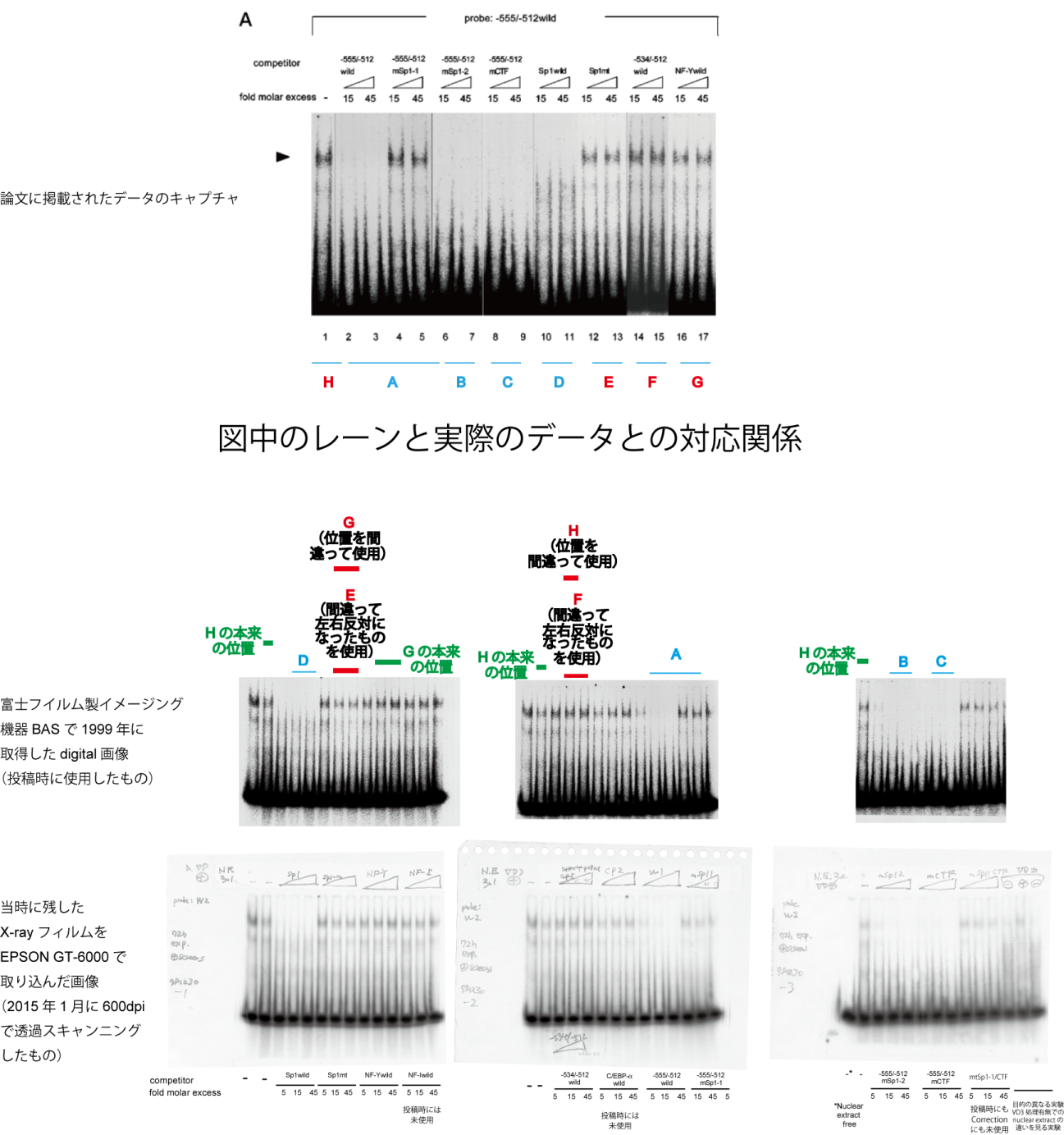
図1: 論文中の図と実際のBAS画像、X-rayフィルムのレーンの対応

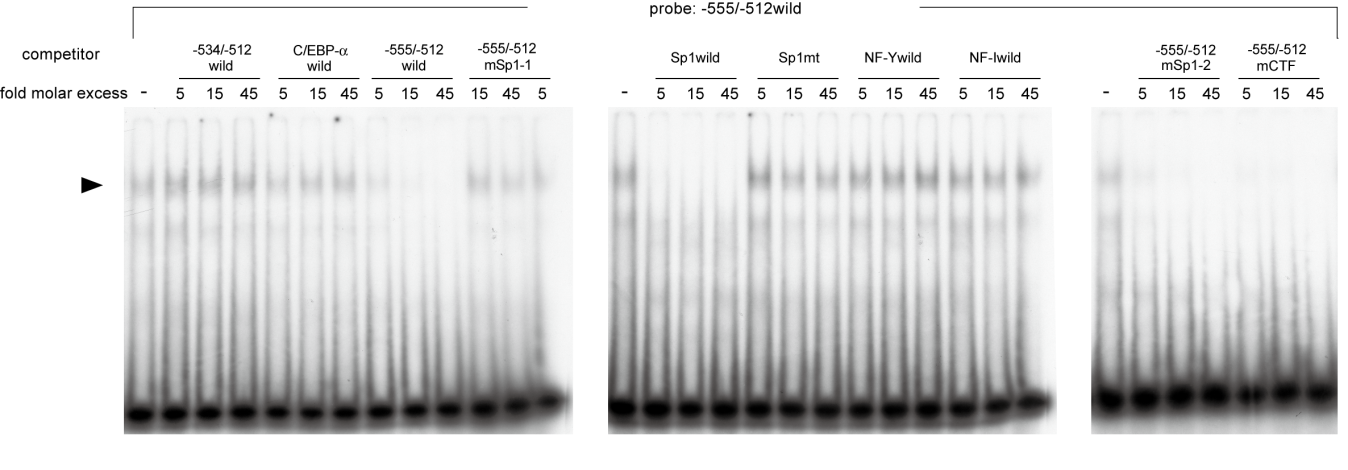
図2: J. Biol. Chem.にCorrectionのために提出した図
上が原図に相当するもの、下がsplitや並び替えを避け、元データのまま作図したもの
